- PLASMIDES ET ÉPISOMES
- PLASMIDES ET ÉPISOMESChaque cellule renferme un ou plusieurs chromosomes dont l’information génétique est portée par une ou plusieurs molécules d’acide désoxyribonucléique. En plus de leurs chromosomes, de nombreuses cellules possèdent d’autres éléments génétiques indépendants, notamment des plasmides. Qu’est-ce qu’un plasmide? Cette définition a évolué au cours du temps en fonction du développement des connaissances scientifiques dans ce domaine. La définition originale de Lederberg en 1952 est génétique: ce sont des déterminants génétiques capables de se transmettre d’une bactérie à l’autre indépendamment du chromosome bactérien; ce sont donc des éléments extrachromosomiques capables d’autoreproduction. Cette définition se basait sur la découverte suivante: l’agent causal du transfert de gènes entre des souches d’Escherichia coli était un plasmide qui pouvait se disséminer dans la population bactérienne. Ensuite, il a été montré chez d’autres entérobactériacées, comme les Shigella , que l’acquisition simultanée de la résistance à plusieurs drogues pouvait être transférée en bloc à d’autres bactéries de la même espèce ou d’espèces différentes et que ces résistances étaient portées par un plasmide ou plusieurs (facteurs R).Ces premiers plasmides, définis par leur fonction, ont été isolés comme des molécules d’ADN circulaires fermées, c’est-à-dire sans extrémité libre (cf. infra , photo). La recherche de telles molécules a été effectuée dans de nombreux organismes, et on a pu ainsi isoler des molécules circulaires d’ADN (fig. 1) à partir de nombreuses bactéries, mais aussi d’eucaryotes. On a pu montrer que ces molécules se répliquaient indépendamment du chromosome et étaient donc bien des plasmides.Parallèlement, Jacob et Wollman ont proposé le terme d’épisomes pour des plasmides pouvant exister soit à l’état autonome libre, soit à l’état intégré dans le chromosome bactérien. À l’état intégré, l’épisome occupe une position définie sur le chromosome bactérien et est répliqué comme une partie de celui-ci. À cette occasion il est apparu que certains virus ou phages pouvaient, dans certaines conditions physiologiques ou après mutation, se comporter comme des plasmides. Le cas ne sera pas envisagé dans ce chapitre (cf. BACTÉRIOPHAGES, LYSOGÉNIE, VIRUS).Certains plasmides atteignent des tailles gigantesques: c’est le cas par exemple des mégaplasmides de Rhizobium qui ont un poids moléculaire supérieur à 450 憐 106 daltons; c’est-à-dire plus de 10 p. 100 de la taille du chromosome. Est-ce encore un plasmide ou un minichromosome?Enfin, chez des levures et des champignons filamenteux et des bactéries du genre Streptomyces , à côté de plasmides circulaires, des plasmides linéaires ont pu être mis en évidence, certains de ces plasmides étant constitués d’ARN et non d’ADN.Il est donc devenu nécessaire d’élargir la définition originale. Un plasmide peut alors être défini comme un élément extrachromosomique constitué soit d’ADN soit d’ARN, capable de se maintenir indépendamment du ou des chromosomes dans la cellule qui l’héberge et celles qui en seront issues. Cependant, à la différence du chromosome, un plasmide n’est pas toujours indispensable à la survie de la cellule, à laquelle il confère néanmoins souvent – mais pas nécessairement – un avantage sélectif. Il peut donc être perdu, ce qui n’est évidemment pas le cas du ou des chromosomes. Au niveau d’un ensemble d’individus tous auront le ou les mêmes chromosomes, mais tous ne renfermeront pas un ou plusieurs plasmides.Enfin, pour un élément génétique donné, l’état plasmidique peut être permanent ou transitoire. Dans ce dernier cas, cet élément peut, par exemple, s’intégrer au génome proprement dit ou exprimer des fonctions virales ou de transposition.1. Diversité des plasmidesSi les plasmides ont d’abord été identifiés chez les entérobactériacées, on considère maintenant qu’ils sont universellement répartis aussi bien chez les procaryotes que chez les eucaryotes.Il serait vain de faire ici la liste de tous les plasmides connus et de leurs hôtes, ce qui serait extrêmement long et fastidieux, et on se bornera à illustrer leurs fonctions et leur répartition par un rapide inventaire.Rôle des plasmides des procaryotesPropriété de transfertLa propriété de transfert implique, par exemple, que le plasmide soit capable de synthétiser des poils spécifiques ou poils sexuels qui vont lui permettre de se transférer et de se propager d’un individu à l’autre, en provoquant éventuellement soit le transfert de gènes chromosomiques (mobilisation), soit celui d’autres plasmides, indépendants, incapables de se transférer par eux-mêmes (cf. infra ).Résistance aux antibiotiques et production de toxinesLes problèmes de résistance des bactéries pathogènes aux antibiotiques, auxquels on est très souvent confronté en médecine humaine et vétérinaire, sont dus, dans la plupart de ces cas, à la présence de plasmides porteurs de facteurs R (R = résistance). Ces facteurs se rencontrent aussi bien chez les bactéries Gram négatif, telles que les colibacilles, que chez les Gram positif, comme le staphylocoque doré. Grâce aux échanges possibles entre individus, cette résistance peut être acquise rapidement par un grand nombre de bactéries.De plus, certains plasmides peuvent porter des gènes responsables de la synthèse de toxines et rendre ainsi la bactérie pathogène. C’est le cas des plasmides «Ent» permettant la synthèse d’entérotoxines chez le colibacille, qui devient alors la cause de diarrhées.Fixation de l’azote atmosphériqueL’azote est, après l’eau, le facteur limitant principal de la production agricole qui, elle-même, conditionne la survie et le développement de l’espèce humaine. La seule réserve d’azote disponible dans le monde est l’azote atmosphérique. Estimée à 4 憐 1015 tonnes, cette réserve est pratiquement infinie, et la majeure partie de l’azote combiné, minéral ou organique de notre planète, provient de la fixation biologique par les bactéries.Par exemple, la symbiose Rhizobium-légumineuses assure près de 50 p. 100 de la fixation annuelle d’azote à la surface du globe. Rhizobium meliloti , symbionte spécifique de la luzerne, renferme plusieurs plasmides, dont un mégaplasmide d’un poids moléculaire de 450 憐 106 daltons environ qui porte l’ensemble des gènes nécessaires à la fixation de l’azote.À propos de ces mégaplasmides, on peut citer celui d’une espèce voisine, Agrobacterium tumefaciens , dont le plasmide Ti est capable, au cours de l’infection d’une plante par la bactérie, de s’intégrer en partie au génome de la plante, ce qui provoque la formation d’un tissu tumoral.Ce type de plasmide a été très étudié, notamment pour son application à la construction de vecteurs pour le génie génétique chez les végétaux.Fonctions cataboliquesUn cas typique est fourni par les plasmides cataboliques de Pseudomonas qui permettent à ces bactéries d’utiliser une grande variété de substrats carbonés comme le camphre (plasmide CAM), le naphtalène (plasmide NaH), le toluène, les xylènes (plasmides XYL), l’octane, l’hexane, le décane (plasmide OCT). Il a été possible de «construire» une bactérie regroupant tous ces plasmides et capable, tout du moins en théorie, de digérer et d’éliminer les nappes d’hydrocarbures [cf. BACTÉRIOLOGIE].Plasmides cryptiquesLes plasmides cryptiques sont ceux pour lesquels aucune fonction n’a pu être mise en évidence.Cas des plasmides des eucaryotesChez la levure Saccharomyces cerevisiae nous avons mis en évidence un plasmide de 2 猪m de longueur qui ne semble coder que pour des fonctions nécessaires à sa maintenance. Ce plasmide est très utilisé comme vecteur de clonage.Plus tard, grâce aux progrès technologiques, des plasmides linéaires ont pu être mis en évidence chez des levures et des champignons filamenteux. Certains de ces plasmides coderaient pour des toxines.2. Propriétés des plasmides bactériensLes plasmides bactériens sont des molécules circulaires constituées d’ADN double brin (bicaténaire) extrachromosomiques, capables de se répliquer de façon indépendante dans les bactéries hôtes et de se transmettre de façon régulière et stable des bactéries mères aux bactéries filles au cours de la croissance bactérienne, bien que leur présence ne soit pas indispensable au métabolisme normal de l’hôte bactérien: on peut isoler des variants dépourvus de plasmide. Cette perte est définitive, la bactérie «guérie» de son plasmide est incapable d’en générer un nouveau, mais elle peut l’acquérir à partir d’une autre bactérie. La présence d’un plasmide dans une souche bactérienne implique deux propriétés fondamentales: la réplication de l’anneau plasmidique, autrement dit la synthèse de matériel plasmidique par la bactérie hôte, et ensuite le transfert de ce matériel, par différentes voies, entre deux bactéries distinctes, comme s’il s’agissait d’un agent contaminant. Des propriétés adaptatives diverses sont liées à ce transfert plasmidique.La réplicationOn appelle réplication l’ensemble des processus qui aboutissent à la synthèse de nouvelles molécules d’ADN plasmidique; identiques au plasmide originel, ces copies plasmidiques seront réparties dans les cellules filles au cours de la division bactérienne. Pour qu’un plasmide soit viable, son génome doit contenir nécessairement l’information pour des fonctions minimales ou fonctions d’autonomie qui définissent son individualité: c’est-à-dire sa réplication autonome, le contrôle de cette réplication et de la répartition de ses copies de façon à maintenir un lien constant avec son hôte bactérien.En 1963, F. Jacob, L. Brenner et F. Cuzin proposent un modèle selon lequel la réplication du plasmide commence en un point précis, l’origine, et progresse d’une manière séquentielle et linéaire vers un autre point spécifique, la terminaison, puis s’arrête; une substance appelée «initiateur», codée par le plasmide lui-même, déclenche la réplication. La fixation du plasmide à la face interne de la membrane cellulaire est indispensable à la réplication et à la répartition des copies. À un certain stade du cycle de développement de la bactérie, un nouveau site de fixation se formerait, déclenchant la réplication, la nouvelle molécule d’ADN s’attachant à ce site. Une cloison entre les deux sites de fixation assure la répartition d’une molécule plasmidique à chaque cellule fille. D’après ce modèle, une très petite partie du génome du plasmide est nécessaire pour assurer les fonctions d’autonomie. L’étude de délétions affectant différents plasmides ainsi que des expériences plus récentes de clonage moléculaire ont permis de confirmer cette hypothèse. Les gènes responsables de l’autonomie, donc de la viabilité du plasmide, sont rassemblés sur ce que l’on appelle une «unité de conduite de réplication», petit segment d’ADN, dont la taille est d’environ 2 000 paires de bases, qui assure la réplication de plasmides cinquante fois plus longs. Actuellement, on définit quatre éléments dans l’unité typique de conduite de la réplication: l’origine (site d’initiation de la réplication), la substance initiatrice, les fonctions génétiques qui contrôlent le nombre de copies, et les fonctions qui assurent la répartition des copies dans les cellules filles. Des études utilisant des techniques de coupure de l’ADN plasmidique par des enzymes, les endonucléases de restriction qui coupent l’ADN en des sites spécifiques, ont permis d’isoler les origines de plusieurs plasmides. L’étude de mutants de plasmides instables à des températures élevées a montré qu’il existe un gène codant pour une protéine diffusible nécessaire à la réplication et que cette protéine initiatrice est spécifique de chaque plasmide. La réplication plasmidique n’est pas comme on l’avait d’abord envisagé couplée à la réplication chromosomique et sous son contrôle. La réplication plasmidique est contrôlée indépendamment de la réplication du chromosome. Le nombre de copies de plasmide par chromosome ou par cellule est très variable selon le plasmide, de une à deux copies à plus d’une centaine de copies; plus le plasmide est grand, et plus le nombre de copies par cellule est faible. Plusieurs plasmides différents peuvent cohabiter dans la même bactérie avec un nombre de copies très différent, le nombre de copies étant déterminé par un système de régulation propre à chacun d’eux. Pour certains plasmides, il a été démontré que le nombre de copies est contrôlé par une ou plusieurs protéines inhibant la réplication; il s’agit là d’un modèle de régulation négative par un répresseur. Pour qu’un plasmide présent dans une bactérie en un nombre limité de copies se transmette de façon stable au cours des générations, un mécanisme assurant l’égale répartition des copies dans les cellules filles doit exister. Des études sur des mutants plasmidiques à initiateur de réplication thermosensible ont apporté la preuve de l’existence de ce mécanisme de répartition. Les bactéries hébergeant de tels mutants plasmidiques peuvent se diviser à 42 0C, mais la réplication du plasmide ne se fait plus. À chaque génération, les copies se répartissent en nombre égal dans les cellules filles, aboutissant au fil des générations à des bactéries porteuses d’une seule copie plasmidique, puis à des bactéries dépourvues de plasmide et sensibles aux antibiotiques auxquelles elles étaient résistantes grâce à la présence du plasmide. À côté de ces fonctions nécessaires à leur existence même, les plasmides peuvent porter des gènes codant pour de très nombreuses propriétés dont certaines ont des conséquences d’une extrême importance pour l’épidémiologie de la résistance aux antibiotiques et pour la pathogénicité des bactéries.Le transfertClassiquement, on divise les plasmides en deux catégories: les plasmides conjugatifs et les plasmides non conjugatifs.Un plasmide conjugatif code pour des fonctions nécessaires à son transfert d’une bactérie A à une bactérie B; il est dit autotransmissible:
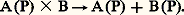 Les plasmides conjugatifs ont une masse moléculaire supérieure à 30 mégadaltons (Md), leur nombre de copies par chromosome est faible (de 1 à 3), leur présence détermine à la surface de la bactérie la synthèse de pili sexuels nécessaire à la conjugaison. Le transfert est le plus souvent réprimé et s’effectue à faible fréquence, de l’ordre de 10 size=1漣4 par bactérie donatrice. La conjugaison est réglée par une unité de transcription composée d’une vingtaine de gènes: l’opéron tra . Son déroulement fait intervenir plusieurs étapes successives: interactions pili-paroi aboutissant à la formation d’agrégats de bactéries, synthèse de l’ADN conjugatif ou «mobilisation», transfert dans la bactérie réceptrice d’un seul brin d’ADN plasmidique, synthèse du brin d’ADN complémentaire dans la bactérie receveuse et circulation de l’ADN plasmidique double brin (fig. 6).Les plasmides non conjugatifs ont une masse moléculaire beaucoup plus petite que les plasmides conjugatifs, le plus souvent autour de 5 Md. Le nombre de copies de ces petits plasmides par copie de chromosome est élevé, supérieur à 10. Ces plasmides ne possèdent pas les gènes nécessaires à la conjugaison, cependant la plupart d’entre eux peuvent assurer l’étape de synthèse de l’ADN conjugatif. Si la bactérie hôte héberge par ailleurs un plasmide autotransférable, au cours de son transfert ce dernier est capable d’assurer le transfert simultané du plasmide non conjugatif par un mécanisme que l’on appelle «mobilisation».En définitive, le transfert et la dissémination des plasmides de bactérie à bactérie peut se faire soit par conjugaison, soit par mobilisation. La conjugaison est un phénomène très répandu chez les bacilles à Gram négatif; plus récemment ce phénomène a été décrit chez les bactéries à Gram positif (streptocoques, staphylocoques, streptomyces, Clostridium ...).Conjugatifs ou non, les plasmides peuvent être disséminés par d’autres mécanismes: ainsi, au cours du transfert par transduction , l’ADN plasmidique est incorporé dans un bactériophage et il est injecté en même temps que l’ADN du phage dans la bactérie réceptrice. Enfin, l’ADN plasmidique peut être acquis par une nouvelle bactérie par un mécanisme de transformation (cf. PARASEXUALITÉ).Les autres fonctions codées par les plasmidesEn plus des gènes qui assurent leur réplication et leur dissémination, les plasmides sont le support d’un grand nombre de déterminants génétiques qui confèrent à l’hôte bactérien des activités biologiques très variées. Les plasmides codant pour la résistance aux antibiotiques ont été les plus étudiés à cause de leur importance clinique. Les plasmides peuvent coder pour la résistance à d’autres agents toxiques pour la bactérie: bactériocines, phages, métaux lourds, détergents, radiations, agents mutagènes, sérum. Les plasmides codant pour des caractères métaboliques confèrent à leurs hôtes la capacité d’utiliser comme aliments de nouvelles substances, leur permettant une meilleure adaptation à l’environnement. Enfin, certains caractères codés par des plasmides jouent un rôle essentiel dans la pathogénicité et la virulence de la bactérie hôte: facteurs d’adhérence, production de toxines, d’hémolysines, capacité d’induire des tumeurs chez les plantes. Un plasmide peut coder, et c’est fréquemment le cas, à la fois pour la résistance aux antibiotiques et une ou plusieurs autres propriétés.Structure et isolementLe plasmide est constitué d’une double hélice d’ADN circulaire surenroulé. Chaque brin est formé d’un enchaînement de quatre bases: adénine (A), guanine (G), thymine (T), cytosine (C) qui se succèdent selon une séquence caractéristique. La séquence des bases d’un brin est complémentaire de la séquence des bases de l’autre brin, A correspondant à T et C à G. La taille des plasmides est très variable, de 1/10 à 1/1 000 de la taille du chromosome bactérien.L’isolement des plasmides peut être réalisé par plusieurs techniques: électrophorèse, ultracentrifugation, microscopie électronique. La microscopie électronique n’est pas un moyen d’isolement mais permet la visualisation et la mesure.Électrophorèse en gel d’agarose d’un lysat bactérienLes bactéries sont lysées, l’ADN est extrait, et un échantillon d’ADN purifié est placé dans un puits à l’une des extrémités de la couche de gel. On fait passer un courant électrique. Les molécules d’ADN plasmidiques migrent vers l’électrode positive à une vitesse inversement proportionnelle au logarithme de leur longueur (fig. 2). Les bandes d’ADN dans le gel sont révélées par un colorant fluorescent qui se fixe électivement sur l’ADN, le bromure d’éthidium, et photographiées en lumière ultraviolette. Cette technique est simple, rapide, permet d’analyser un grand nombre de souches en même temps et a un grand intérêt épidémiologique. Cependant les résultats obtenus par cette technique sont difficilement interprétables lorsque la bactérie héberge plusieurs plasmides de résistance différents, ce qui est fréquent chez les souches résistantes isolées en milieu hospitalier. En effet, chaque plasmide peut donner naissance à plusieurs bandes dans le gel selon sa conformation: structure double brin surenroulée, forme circulaire ouverte par coupure d’un des brins de l’ADN, enfin forme linéaire correspondant à la coupure des deux brins.Ultracentrifugation en chlorure de césium-bromure d’éthidiumIl est possible d’identifier l’ADN plasmidique et de le séparer de l’ADN chromosomique par centrifugation à très grande vitesse dans un gradient de densité de chlorure de césium. L’ADN plasmidique surenroulé est capable d’intercaler entre ses bases moins de bromure d’éthidium que l’ADN chromosomique linéaire. Cette fixation différentielle crée une différence de densité aboutissant au cours de la centrifugation à la formation dans le gradient de deux bandes séparées correspondant l’une à l’ADN chromosomique, l’autre à l’ADN plasmidique (fig. 3). L’ADN plasmidique ainsi purifié peut être conservé et analysé plus avant par microscopie électronique.Microscopie électroniqueL’examen au microscope électronique d’un étalement du contenu plasmidique d’une souche bactérienne permet de déterminer le nombre de plasmides hébergés par cette souche et de mesurer la taille de chacun des plasmides (fig. 1).Critères d’identification et classificationIncompatibilitéDeux plasmides identiques ou étroitement apparentés ne peuvent pas coexister de façon stable dans la même bactérie: ils sont dits incompatibles. À partir d’une bactérie dans laquelle on introduit deux plasmides apparentés, au fur et à mesure de la succession des générations, les bactéries filles se divisent en deux populations qui ne possèdent chacune que l’un ou l’autre plasmide. Au contraire, deux plasmides non apparentés peuvent se maintenir de façon stable dans la même bactérie: ils sont dits compatibles. On isole couramment de bactéries multirésistantes six à huit plasmides différents coexistant dans la même bactérie donc compatibles. On définit ainsi des groupes d’incompatibilité. Deux plasmides incompatibles appartiennent au même groupe d’incompatibilité, et deux plasmides compatibles appartiennent à des groupes d’incompatibilité différents. L’incompatibilité est un phénomène que l’on peut observer dans tous les genres bactériens dont on a étudié les plasmides. Cependant, les plasmides d’entérobactéries sont ceux qui ont été les plus étudiés, on distingue une vingtaine de groupes d’incompatibilité différents chez les plasmides d’entérobactéries autotransférables chez Escherichia coli. Les plasmides du même groupe d’incompatibilité ont des tailles comparables, un degré élevé d’homologie entre leurs ADN (cf. infra ), une spécificité d’hôte voisine. En revanche, des plasmides appartenant à des groupes d’incompatibilité différents peuvent porter des caractères de résistance aux mêmes antibiotiques (cf. chap. Variation ), et il n’existe pas de relation entre groupe d’incompatibilité et caractères de résistance codés par les différents plasmides. Le phénomène d’incompatibilité est étroitement lié à la réplication et à la répartition des plasmides; il semble que des plasmides du même groupe d’incompatibilité possèdent des unités de conduite de réplication identiques, et le même appareil de séparation. Il existerait donc au moins autant d’unités de réplication et de systèmes de répartition que de groupes d’incompatibilité.Spectre de digestion de l’ADN plasmidique par des endonucléases de restrictionLes endonucléases de restriction sont des enzymes d’origine bactérienne qui coupent l’ADN plasmidique en des sites spécifiques, chacune de ces enzymes reconnaissant une séquence de paires de base particulière. La coupure du plasmide par différentes enzymes de restriction aboutit pour chaque enzyme à une série de fragments d’ADN; les fragments obtenus sous l’action d’une endonucléase donnée sont séparés par électrophorèse en gel d’agarose selon leur taille. Chaque sorte de plasmide donne ainsi, après traitement par une endonucléase, un diagramme de coupure caractéristique. Deux plasmides ayant des diagrammes de coupure par plusieurs enzymes superposables appartiennent à des espèces moléculaires voisines ou identiques (fig. 4).HybridationLa parenté entre les ADN de deux plasmides peut être mesurée par hybridation; il peut s’agir d’hybridation entre plasmides entiers ou entre fragments d’ADN plasmidique après coupure par une endonucléase de restriction. L’ADN non radioactif du premier plasmide est fixé soit entier, soit après fragmentation et séparation par électrophorèse, sur un filtre, et l’hybridation se fait avec l’ADN radioactif préparé avec l’autre plasmide. La mesure de la radioactivité fixée sur le filtre se fait soit par comptage de la radioactivité dans le cas des plasmides entiers, soit par autoradiographie pour les ADN fragmentés. Les plasmides appartenant au même groupe d’incompatibilité ont un très fort pourcentage d’homologie ADN-ADN.Analyse d’hétéroduplex en microscopie électroniqueOn peut mesurer l’importance de l’homologie entre les ADN de deux plasmides par l’analyse d’hétéroduplex en microscopie électronique. Un hétéroduplex est le résultat de la réassociation de deux brins d’ADN provenant chacun d’un plasmide différent. Les régions constituées de doubles brins d’ADN correspondent aux régions d’homologie. Les boucles simple brin correspondent aux régions hétérologues. Cette technique permet de détecter et de mesurer l’homologie entre deux plasmides (fig. 5).3. Plasmides R: plasmides de résistance aux antibiotiquesHistoriqueC’est vers la fin des années cinquante que deux microbiologistes japonais, Ochiai et Akiba, observèrent au cours d’une épidémie de dysenterie bacillaire l’apparition de bactéries résistantes à trois antibiotiques à la fois, rendant les malades atteints insensibles au traitement par des antibiotiques habituellement efficaces. Un mécanisme de mutation chromosomique-sélection, décrit par Ledeberg en 1940 et qui permettait d’expliquer les résistances bactériennes observées jusque-là, ne permettait pas d’expliquer cette triple résistance simultanée à trois familles d’antibiotiques différentes. Les auteurs japonais montrèrent alors que cette résistance multiple se transférait entre bactéries intestinales dans le tube digestif des malades, puis ils reproduisirent ce transfert in vitro entre colibacilles et shigelles et démontrèrent que ces bactéries résistantes possèdent en plus de leur chromosome d’autres structures composées d’ADN portant les gènes responsables de cette résistance multiple transférable: ce sont les plasmides de résistance aux antibiotiques ou plasmides R. La découverte de ces éléments génétiques extrachromosomiques mobiles apparut d’emblée d’une importance considérable en clinique par leur incidence sur l’arsenal thérapeutique antibiotique. Les plasmides R permettent aux bactéries d’acquérir la résistance à un ou plusieurs antibiotiques d’emblée; cette résistance est transférable de bactérie à bactérie de la même espèce mais aussi d’espèces voire de genres différents; la résistance peut apparaître ainsi dans une espèce bactérienne jusque-là sensible, la sélection par un antibiotique peut être responsable de la sélection de la résistance à plusieurs antibiotiques non employés, enfin la coexistence possible de plusieurs plasmides R dans une seule cellule bactérienne permet d’expliquer l’émergence dans les milieux à forte pression de sélection antibiotique comme le milieu hospitalier de bactéries multirésistantes responsables d’échec thérapeutique. Depuis leur découverte chez les entérobactéries, des plasmides R ont pu être mis en évidence chez la plupart des bactéries d’intérêt médical, vétérinaire ou industriel.Espèces bactériennes hébergeant des plasmidesDepuis leur découverte chez Shigella dysenteriae , des plasmides de résistance ont été isolés dans la plupart des bactéries pathogènes. Des plasmides ont été mis en évidence chez les bacilles à Gram négatif (entérobactéries diverses, Yersinia , Pseudomonas , Acinetobacter , Aeromonas , Vibrio , Haemophilus , Pasteurella , Bordetella , Campylobacter , Bacteroides fragilis ), les bacilles à Gram positif (Actinomyces , Bacillus , Clostridium perfringens ), les cocci à Gram positif (staphylocoques, streptocoques) et les cocci à Gram négatif (gonocoques). Le pneumocoque et le méningocoque restent les deux seules bactéries de grande importance en pathologie infectieuse dépourvues pour l’instant de plasmides. Des plasmides de résistance peuvent être isolés de nombreuses bactéries de l’environnement, en particulier chez les Streptomyces , bactéries productrices d’antibiotiques. Ces bactéries possèdent les gènes de résistance qui les protègent de l’action de l’antibiotique qu’elles produisent. Ces bactéries productrices d’antibiotiques seraient le réservoir et l’origine de la dissémination des gènes de résistance chez les bactéries pathogènes.DiversificationDeux mécanismes, la mutation et la transposition, permettent la diversification du matériel plasmidique.MutationDes mutations spontanées ou provoquées par des agents mutagènes permettent d’isoler à partir du plasmide originel des variants ou délétants qui sont très utiles pour étudier les propriétés des plasmides: étude de la réplication à l’aide de mutants thermosensibles, étude du transfert avec des mutants déréprimés pour le transfert ou au contraire mutants ayant perdu la transférabilité. Ces mutants ont peu d’incidence sur le phénomène de résistance des antibiotiques.TranspositionLes plasmides peuvent acquérir de nouveaux gènes, en particulier des gènes de résistance aux antibiotiques par le mécanisme de transposition. Certains gènes responsables de la résistance aux antibiotiques sont inclus dans un petit fragment d’ADN qui a la propriété de se détacher ou de «s’exciser» de la molécule d’ADN qui le contient et de s’insérer sur une autre molécule d’ADN. Ces morceaux d’ADN sauteurs ont été appelés «transposons». Certains gènes de résistance sont donc capables par ce mécanisme de sauter d’un plasmide à un autre, d’un plasmide sur un chromosome ou d’un chromosome sur un plasmide. Le phénomène de transposition est un mécanisme qui assure une dissémination très efficace des gènes de résistance. La transposition est un phénomène différent de la recombinaison classique. Elle ne nécessite pas l’enzyme recombinase, elle ne nécessite pas d’homologie entre les ADN donneurs et receveurs du transposon. C’est ainsi que des plasmides appartenant à des groupes d’incompatibilité différents et sans homologie ADN-ADN peuvent porter des gènes de résistance identiques. Les transposons connus mesurent de 2,5 à 20 kilobases, et portent en plus des gènes responsables de la résistance aux antibiotiques, des gènes codant pour des enzymes responsables du mécanisme de transposition et de sa régulation. De nombreux transposons codant pour la résistance à différents antibiotiques ont été caractérisés: 廓-lactamines, streptomycine, kanamycine, gentamicine, tétracycline, érythromycine, chloramphénicol, triméthoprime, sulfamides, phosphomycine. La transposition permet d’expliquer d’une part comment des plasmides R sont capables au cours de leur évolution d’acquérir de nouveaux gènes de résistance; d’autre part elle explique comment des plasmides sans fonction connue et que l’on appelle «cryptiques» peuvent acquérir des gènes de résistance par le mécanisme de transposition et devenir ainsi des plasmides de résistance. La résistance peut apparaître ainsi dans des espèces bactériennes jusque-là sensibles. Ce phénomène a permis d’expliquer l’apparition voici quelques années de souches résistant à la pénicilline chez les Haemophilus et les gonocoques, espèces jusque-là sensibles à cet antibiotique, par la transposition sur des plasmides cryptiques, connus dans ces espèces, de gènes de résistance provenant d’Escherichia coli .Importance pratiqueLes mécanismes de résistance codés par les plasmidesLes plasmides sont capables de rendre les bactéries résistantes par différents mécanismes qui impliquent généralement la synthèse de protéines codées par des gènes plasmidiques: production d’enzymes inactivant l’antibiotique en modifiant sa molécule, modification de la cible de l’antibiotique par une enzyme, diminution de la quantité d’antibiotique transportée à l’intérieur de la bactérie, substitution d’une molécule nouvelle à la place de la cible de l’antibiotique. La majorité des antibiotiques est inactivée par l’un ou l’autre de ces mécanismes, et rares sont les antibiotiques pour lesquels aucune résistance plasmidique n’a été décelée.Outils pour génie génétiqueL’existence de plasmides de résistance n’a pas que des aspects néfastes en amputant l’arsenal thérapeutique. Ce sont des outils particulièrement efficaces dans les expériences de génie génétique. Un des champs d’application privilégiés du génie génétique est l’introduction de gènes codant pour des protéines animales ou humaines dans Escherichia coli pour transformer cette bactérie en usine à fabriquer des protéines d’intérêt thérapeutique ou industriel: hormones, insuline, interféron, etc. Il n’est pas possible actuellement d’introduire directement un fragment d’ADN venant d’organismes supérieurs dans une bactérie et de le fixer sur le chromosome bactérien pour que cette information soit répliquée et transmise à la descendance de la bactérie; un intermédiaire que l’on appelle «vecteur» est indispensable à cette opération. Les plasmides de résistance sont avec les phages les vecteurs essentiellement utilisés. Le plasmide choisi est isolé de la bactérie résistante, purifié, et coupé par une endonucléase de restriction. Le fragment d’ADN étranger peut alors être inséré dans le plasmide ouvert, la molécule hybride recircularisée (fragment d’ADN + plasmide) est introduite chez Escherichia coli. Les bactéries ayant reçu la molécule hybride sont aisément sélectionnées par leur résistance à un antibiotique, codée par le plasmide. Introduire un gène étranger par cet artifice chez Escherichia coli est une opération simple aujourd’hui. Cependant, l’expression de ce gène «importé» dans un environnement qui lui est totalement étranger reste largement imprévisible. Ce phénomène est à l’origine des échecs relativement nombreux rencontrés par les précurseurs dans ce domaine, difficultés qui sont actuellement partiellement résolues.Problèmes épidémiologiquesActuellement, la résistance d’origine plasmidique rend compte de plus de 80 p. 100 de la résistance bactérienne aux antibiotiques. Dans la nature, chez l’homme sain ou malade, et chez les animaux, les bactéries porteuses de plasmides de résistance sont de plus en plus nombreuses. Les quantités considérables d’antibiotiques déversées dans l’environnement à des fins thérapeutiques ou autres depuis quarante ans sont vraisemblablement à l’origine de la sélection de bactéries résistantes. L’antibiosupplémentation de l’alimentation du bétail, en particulier, a été mise en cause dans l’augmentation de fréquence des bactéries multirésistantes. Ainsi, on a pu montré que 100 p. 100 des porcs et des veaux recevant des antibiotiques hébergeaient dans leur tube digestif des entérobactéries porteuses de plasmides de résistance transférables. Chez les éleveurs ne recevant pas d’antibiotiques, mais en contact avec les animaux, le portage digestif d’entérobactéries résistantes est de 50 p. 100 ou plus; en revanche, l’étude d’une population isolée de Bornéo sans contact avec les antibiotiques a montré que moins de 1 p. 100 des bactéries des selles pouvaient transférer leur résistance. Dans l’environnement, on peut isoler des bactéries multirésistantes dans les égouts, sur les plages par exemple. On peut donc supposer que c’est l’utilisation massive des antibiotiques qui est responsable d’un si brutal changement écologique. La dissémination de la résistance s’effectue par plusieurs mécanismes parfois étroitement intriqués. Une souche d’une bactérie pathogène peut acquérir un plasmide de résistance provenant d’une entérobactérie du tube digestif, puis cette souche disséminer dans la population (ou de malade à malade), réalisant de la sorte une épidémie due à une seule et unique souche. Il existe de nombreux exemples de telles épidémies: épidémie de fièvre typhoïde en Amérique latine due à une Salmonella porteuse d’un plasmide de résistance au chloramphénicol et à l’ampicilline, épidémies hospitalières à bactéries multirésistantes responsables d’infections nosocomiales dans un milieu à forte pression de sélection par les antibiotiques. En milieu hospitalier, on peut suivre un autre mode de dissémination de la résistance: des épidémies de plasmides, le même plasmide de résistance se propageant au fil du temps dans plusieurs espèces bactériennes différentes dans le même hôpital. De telles épidémies de plasmides de résistance à la gentamicine et au triméthoprime ont été bien individualisées par la comparaison fine des plasmides isolés des différentes espèces de bactéries résistant aux mêmes antibiotiques. Enfin, plus récemment, on a pu démontrer l’identité des gènes de résistance et isoler des transposons identiques sur différents plasmides hébergés par des espèces ou des genres bactériens très divers. Il s’agit alors d’épidémies de transposons. Une appréciation correcte du danger de la résistance plasmidique repose sur l’épidémiologie des plasmides et des gènes de résistance permettant de connaître, de suivre, éventuellement de prévoir et d’empêcher leur dissémination.La détection de nouvelles résistances et la surveillance de l’évolution des résistances connues nécessitent une étroite collaboration entre les thérapeutes, les microbiologistes, les épidémiologistes et les biologistes cellulaires. Enfin, il existe un lien indiscutable entre la résistance des bactéries et l’utilisation des antibiotiques dans un hôpital; une meilleure connaissance de l’écologie locale de la résistance et des différences notables qui existent dans le degré du pouvoir sélectionnant de chaque antibiotique devrait conduire à des modifications dans les habitudes de prescription et à une diminution de la résistance.4. Facteurs sexuels bactériensLe facteur sexuel F d’Escherichia coli est un épisome dont la taille est d’environ 1 p. 100 de celle du chromosome et dont la propriété essentielle est de rendre «fertiles» les bactéries qui le portent. Cette fertilité se manifeste de différentes manières suivant que le facteur sexuel est autonome (bactérie +) ou intégré dans le chromosome (bactérie Hfr). Les bactéries + ou Hfr sont dites donatrices ou mâles, alors que les bactéries dépourvues de facteur sexuel (F size=1漣) sont dites réceptrices ou femelles.La recombinaison par conjugaison bactérienneLa découverte du facteur F est liée à l’étude de la recombinaison génétique chez Escherichia coli K12. En 1946, Lederberg et Tatum montraient que, en mélangeant deux cultures bactériennes X (abc DEF) et Y (ABC def ) respectivement polyauxotrophes, c’est-à-dire exigeantes en produits variés (abc pour X, et def pour Y), on récupérait des bactéries prototrophes Z (ABCDEF) ne nécessitant aucun de ces produits. Les bactéries Z, ne pouvant être des mutants réverses de X ou Y en raison du changement simultané de plusieurs caractères, sont des «recombinants» entre X et Y. Rapidement, on s’aperçut que les bactéries X et Y ne jouaient pas un rôle identique. La survie d’une des deux bactéries X ou Y est nécessaire à la production de recombinants, alors que l’on peut tuer l’autre avant ou pendant le croisement sans diminuer le nombre de recombinants qui apparaissent. La bactérie Y, par exemple, dont la survie est absolument nécessaire, est la bactérie réceptrice: en elle a lieu le processus de recombinaison au niveau moléculaire. La bactérie X est alors la bactérie donatrice (F+). À partir des bactéries +, on a pu isoler des variants qui ne donnent plus de recombinants avec une souche F size=1漣. Par ailleurs, on peut classer les souches d’Escherichia coli en deux types: les souches qui, croisées entre elles, ne donnent jamais de recombinants (souche F size=1漣) et les souches qui donnent des recombinants avec n’importe quelle autre souche (souche fertile +).Les types sexuels + et F size=1漣Le caractère + ou F size=1漣 qui donne aux bactéries le phénotype donneur ou receveur apparaissait donc jusqu’en 1953 comme un caractère génétique classique, déterminé par le chromosome et qui pouvait muter par exemple de + vers F size=1漣. Cependant, certaines propriétés très particulières du caractère + ont montré qu’il n’était pas codé par des gènes chromosomiques. Lors d’un croisement entre une souche + et une souche F size=1漣, on obtenait de l’ordre de 10 size=1漣6 à 10 size=1漣7 recombinant pour les caractères chromosomiques; en revanche, la majorité des bactéries réceptrices sont converties en bactéries +. C’était la première preuve d’indépendance du facteur F par rapport au chromosome. Cette indépendance devait se confirmer par le fait que, lorsqu’on mélange un nombre connu de bactéries donatrices avec un nombre plus grand de bactéries F size=1漣, ces dernières sont converties en bactéries + à une vitesse supérieure à celle de la multiplication bactérienne. Cela signifie que le facteur F se réplique de manière autonome et à une vitesse qui peut être supérieure à celle du chromosome, ce qui lui permet d’envahir par transfert toute une culture: on dit qu’il est «infectieux» (fig. 6). Enfin, le facteur F peut être spontanément perdu par une souche, à un taux qui est en général beaucoup plus grand que le taux de mutation. F est donc un élément indépendant du chromosome, capable de se répliquer à une vitesse propre et de se transférer de bactérie à bactérie par conjugaison à haute fréquence. C’est un plasmide autotransférable.Les mutants HfrÀ partir de bactéries +, Cavalli-Sforza et Hayes ont isolé des mutants qui produisaient mille fois plus de recombinants lorsqu’on les croisait avec des bactéries F size=1漣. Ces mutants se comportaient comme des «supermâles»: on les a appelés souches Hfr (haute fréquence de recombinaison). En plus du problème de la fréquence des recombinants, les croisements Hfr 憐 F size=1漣 présentaient essentiellement deux caractères qui les distinguaient des croisements + 憐 F size=1漣. Les croisements + 憐 F size=1漣 conduisent à un nombre faible mais constant de recombinants Z (ABCDEF), quel que soit le caractère sélectionné. Cela montre donc que les bactéries donatrices + peuvent transférer n’importe quel segment de leur chromosome avec une égale fréquence. Au contraire, dans un croisement Hfr 憐F size=1漣, le nombre de recombinants formés est très inégal suivant le caractère sélectionné. Par ailleurs, sauf cas exceptionnel, les bactéries réceptrices ne sont pas transformées en bactéries + dans un croisement Hfr 憐 F size=1漣. Ces propriétés des donneurs Hfr ont suggéré que, dans ces bactéries, le facteur sexuel F existe sous un état différent de celui sous lequel il existe dans des bactéries +. Il a été démontré par la suite que ce second état du facteur F est l’état intégré au chromosome bactérien où l’ADN de F est en continuité avec l’ADN de la cellule hôte (fig. 7). Nous avons déjà signalé qu’on a donné le nom d’épisome à de tels éléments génétiques capables d’exister sous deux états. Cette généralisation, principalement due à Jacob et Wollman, est née de l’étude du facteur F mais aussi des bactériophages tempérés et des facteurs colicinogènes.Équilibre réplicatif et notion de répliconJacob, Brenner et Cuzin ont proposé d’appeler «réplicon» les structures génétiques dont la réplication autonome est indépendante de celle du chromosome. Un réplicon est donc une unité de réplication. Dans cette hypothèse, que des travaux ultérieurs ont partiellement confirmée, un réplicon a au moins deux types de fonctions et comprend deux sortes de matériel génétique au sens fonctionnel.Le premier type comporte les fonctions obligatoires intervenant spécifiquement dans la réplication et permettant à la molécule d’ADN qui les porte de se répliquer avec une vitesse et une régulation caractéristiques de ce replicon. Les déterminants génétiques impliqués dans cette fonction seraient au moins deux. Il existerait un gène de structure codant pour la synthèse d’une protéine dite initiatrice qui agirait sur un second gène, le réplicateur, «véritable promoteur de la réplication». Lorsque la protéine initiatrice agit sur le réplicateur, la réplication est amorcée et se poursuit tout le long de la molécule d’ADN adjacente au réplicateur (position cis). Ces deux gènes sont absolument spécifiques et l’initiateur produit par un réplicon donné, par exemple un phage, reconnaît le réplicateur de ce phage, mais pas le réplicateur du chromosome bactérien ou d’un autre type de phage.Le second type de fonction est variable et caractériel le type de matériel génétique que l’on étudie: le facteur F porte des gènes qui permettent un transfert de matériel génétique; les facteurs de résistance (cf. chap. 3) codent pour des enzymes qui détruisent certains antibiotiques; le chromosome bactérien contient toute l’information nécessaire à la multiplication de la bactérie. Ces gènes se répliquent à la vitesse du réplicon qui les porte.On sait actuellement que le facteur F se multiplie indépendamment du chromosome mais en accord avec lui de façon à conserver le rapport de un épisome par chromosome. Cependant, il peut se produire des cas où cet équilibre est rompu; par exemple, si des bactéries + sont mises en contact avec des bactéries F size=1漣, le facteur F envahit la culture par le biais du transfert (cf. chap. 2, Le Transfert ). Par ailleurs, certaines bactéries peuvent perdre le facteur F et deviennent F size=1漣. La fréquence de cette perte est de l’ordre de 10 size=1漣2 à 10 size=1漣4 selon les souches; elle peut être accrue si on traite les bactéries par des colorants comme l’acridine orange. Notons que la transition spontanée du caractère + vers F size=1漣 est possible alors que le passage de l’état F size=1漣 vers l’état + nécessite le contact avec une souche +.La réplication du facteur F a été étudiée en utilisant des mutants thermosensibles. Dans ces expériences, on utilise en général des facteurs F (cf. infra ) qui portent des marqueurs génétiques faciles à repérer comme le caractère lac+ (utilisation du lactose). Si le facteur F size=1漣 lac+ est étudié dans une bactérie lac size=1漣 (incapable d’utiliser le lactose), le caractère lac+ sera un témoin de la présence du facteur F . Il a été obtenu des mutants thermosensibles de F lac+ que l’on désigne Fts-lac+. Dans ces bactéries, le Fts-lac+ se réplique à basse température (25 0C), mais à haute température (42 0C) les bactéries deviennent lac size=1漣 car le Fts-lac+ ne se multiplie pas. À 42 0C, on trouve cependant quelques clones lac+: dans ces bactéries, le Fts-lac+ a donc pu se perpétuer. Une étude approfondie de ces bactéries a montré qu’elles ont conservé l’épisome dont la réplication est thermosensible, mais que celui-ci s’est intégré dans le chromosome, et est donc répliqué par le «réplicon chromosome». Chez ces mutants, le chromosome «sauve» l’épisome de l’effet de la température. De manière réciproque, des mutants de chromosomes bactériens thermosensibles pour la réplication peuvent être sauvés par l’intégration d’un facteur F normal. C’est ici le facteur F qui prend la réplication du chromosome sous son contrôle. L’ensemble de ces expériences illustre précisément les prévisions que l’on pouvait faire à partir de la théorie du réplicon.Propriétés des bactéries +Les propriétés particulières qui différencient les bactéries portant le facteur F des bactéries F size=1漣 dépourvues de ce facteur sont liées à l’expression de la fonction de transfert du facteur sexuel. Tout d’abord, les bactéries + présentent des différences morphologiques liées essentiellement à l’existence à leur surface d’un petit nombre de «poils» spécifiques appelés «F pili». La synthèse des F pili est constante chez les bactéries +, alors que la synthèse d’éléments semblables est soumise à régulation chez des bactéries portant des facteurs de résistance. Si l’on enlève les F pili par agitation de la culture dans un mixer, ou si l’on utilise un mutant dénué de pili, le transfert génétique du facteur F n’a pas lieu. Comme ils sont creux, ces «poils» sont peut-être des organes de copulation à travers lesquels s’effectue le transfert de matériel génétique. Ce point a été discuté car certains auteurs estiment que les pili sont simplement des «câbles d’amarrage» servant durant la période initiale de la conjugaison à l’appariement des bactéries. L’existence de ces poils explique un certain nombre de différences biochimiques et immunologiques entre les bactéries donatrices et réceptrices. Par ailleurs, ces F pili portent les récepteurs de bactériophages qui ne se fixent et ne se multiplient que chez les bactéries mâles.Sur le plan biochimique, on peut retrouver dans le cytoplasme des bactéries + des molécules d’ADN circulaire ayant sensiblement la même composition en bases nucléiques que l’ADN du chromosome d’Escherichia coli (50 p. 100 de guanine-cytosine).Au niveau génétique, la propriété de transfert doit donc impliquer l’existence de deux types de gènes: ceux qui conduisent à la synthèse des F pili et ceux qui permettent à la bactérie donatrice d’injecter le facteur F à la bactérie réceptrice tout en restant elle-même +. Des mutants du facteur F ont été isolés, qui sont incapables d’assurer leur propre transfert et le transfert de gènes chromosomiques. La plupart de ces mutants sont incapables de synthétiser des F pili; les autres seraient incapables d’injecter l’ADN dans les bactéries F size=1漣. Il semble y avoir indépendance entre la réplication du facteur F et le transfert de l’ADN entre bactéries conjugantes. Lorsque le facteur F est normal, les bactéries + transmettent le facteur F avec une fréquence élevée et les gènes chromosomiques avec une fréquence faible. On admet que le transfert à basse fréquence des gènes chromosomiques est dû à l’existence dans les cultures de souches + de rares variants Hfr.Propriétés des bactéries Hfr.Les bactéries Hfr présentent les mêmes caractères morphologiques que les bactéries +. Mais les bactéries expriment la fonction de transfert d’une façon différente, liée au fait que le facteur F est intégré dans le chromosome bactérien. Dans ce cas, F est répliqué passivement comme un segment d’ADN chromosomique, mais en revanche «impose» la fonction de transfert à tout le chromosome bactérien. Cela se traduit par les caractères particuliers que présente la conjugaison entre une bactérie Hfr et une bactérie F size=1漣: ces croisements produisent beaucoup plus de recombinants qu’un croisement + 憐 F size=1漣; la proportion des recombinants est très variable suivant le caractère chromosomique sélectionné (de 10 size=1漣3 à 10 size=1漣4); la nature des marqueurs transmis à haute fréquence varie avec l’origine de la source Hfr utilisée. En ce qui concerne le facteur sexuel, il n’est pas infectieux dans les souches Hfr, et il est très rarement transmis lors du processus de conjugaison. Jacob et Wollman ont démontré que le transfert des gènes chromosomiques est orienté et progressif. Ces conclusions reposent sur une étude cinétique du processus dans des expériences de conjugaison interrompue.On peut donner actuellement une explication assez claire du comportement des bactéries Hfr. Les bactéries Hfr sont des mutants de bactéries + dans lesquelles le facteur F s’est intégré dans le chromosome. Le facteur F est alors capable de promouvoir le transfert de gènes chromosomiques. Le lieu d’insertion du facteur F dans ce chromosome détermine quels seront les marqueurs transmis en premier ou en dernier à partir d’une bactérie Hfr donnée (fig. 8). Le nombre de points d’insertion est élevé mais limité, aussi existe-t-il un nombre limité de types d’Hfr. D’autre part, le chromosome bactérien étant cent fois plus long que le facteur F, le transfert du chromosome est long, peut s’interrompre spontanément par cassure de ce chromosome ou dissociation des paires de bactéries. Cela explique que les gènes proches de la «tête» du chromosome de l’Hfr entreront souvent dans la femelle et donneront de nombreux recombinants, alors que les gènes situés en «queue» seront souvent non transférés. D’autre part, dans les souches Hfr, la réplication du facteur F est liée à celle du chromosome, et ce F ne peut donc être infectieux, comme c’est le cas dans les souches +.L’état Hfr est assez instable, et on peut revenir vers l’état + par excision du facteur F. Lors de ce processus, il peut se produire diverses anomalies. Cela conduit alors à la formation d’épisomes portant des fragments de chromosome (fig. 9): ce sont les facteurs F déjà cités. Ils conservent en général toutes les caractéristiques de F, mais emmènent des segments d’ADN bactériens de tailles variables, pouvant atteindre 10 p. 100 du chromosome. Des F peuvent donc transporter des gènes bactériens d’une souche à une autre. Ce phénomène s’appelle la sexduction. Les facteurs F sont très utilisés en génétique bactérienne soit en tant que marqueurs des facteurs F (cf. supra ), soit pour construire des diploïdes partiels (mérodiploïdes). Les bactéries sont en effet haploïdes; et, pour étudier le caractère dominant ou récessif d’une mutation, il faut avoir recours à des artifices génétiques. Les facteurs F et F sont donc des outils très utiles en génétique moléculaire.
Les plasmides conjugatifs ont une masse moléculaire supérieure à 30 mégadaltons (Md), leur nombre de copies par chromosome est faible (de 1 à 3), leur présence détermine à la surface de la bactérie la synthèse de pili sexuels nécessaire à la conjugaison. Le transfert est le plus souvent réprimé et s’effectue à faible fréquence, de l’ordre de 10 size=1漣4 par bactérie donatrice. La conjugaison est réglée par une unité de transcription composée d’une vingtaine de gènes: l’opéron tra . Son déroulement fait intervenir plusieurs étapes successives: interactions pili-paroi aboutissant à la formation d’agrégats de bactéries, synthèse de l’ADN conjugatif ou «mobilisation», transfert dans la bactérie réceptrice d’un seul brin d’ADN plasmidique, synthèse du brin d’ADN complémentaire dans la bactérie receveuse et circulation de l’ADN plasmidique double brin (fig. 6).Les plasmides non conjugatifs ont une masse moléculaire beaucoup plus petite que les plasmides conjugatifs, le plus souvent autour de 5 Md. Le nombre de copies de ces petits plasmides par copie de chromosome est élevé, supérieur à 10. Ces plasmides ne possèdent pas les gènes nécessaires à la conjugaison, cependant la plupart d’entre eux peuvent assurer l’étape de synthèse de l’ADN conjugatif. Si la bactérie hôte héberge par ailleurs un plasmide autotransférable, au cours de son transfert ce dernier est capable d’assurer le transfert simultané du plasmide non conjugatif par un mécanisme que l’on appelle «mobilisation».En définitive, le transfert et la dissémination des plasmides de bactérie à bactérie peut se faire soit par conjugaison, soit par mobilisation. La conjugaison est un phénomène très répandu chez les bacilles à Gram négatif; plus récemment ce phénomène a été décrit chez les bactéries à Gram positif (streptocoques, staphylocoques, streptomyces, Clostridium ...).Conjugatifs ou non, les plasmides peuvent être disséminés par d’autres mécanismes: ainsi, au cours du transfert par transduction , l’ADN plasmidique est incorporé dans un bactériophage et il est injecté en même temps que l’ADN du phage dans la bactérie réceptrice. Enfin, l’ADN plasmidique peut être acquis par une nouvelle bactérie par un mécanisme de transformation (cf. PARASEXUALITÉ).Les autres fonctions codées par les plasmidesEn plus des gènes qui assurent leur réplication et leur dissémination, les plasmides sont le support d’un grand nombre de déterminants génétiques qui confèrent à l’hôte bactérien des activités biologiques très variées. Les plasmides codant pour la résistance aux antibiotiques ont été les plus étudiés à cause de leur importance clinique. Les plasmides peuvent coder pour la résistance à d’autres agents toxiques pour la bactérie: bactériocines, phages, métaux lourds, détergents, radiations, agents mutagènes, sérum. Les plasmides codant pour des caractères métaboliques confèrent à leurs hôtes la capacité d’utiliser comme aliments de nouvelles substances, leur permettant une meilleure adaptation à l’environnement. Enfin, certains caractères codés par des plasmides jouent un rôle essentiel dans la pathogénicité et la virulence de la bactérie hôte: facteurs d’adhérence, production de toxines, d’hémolysines, capacité d’induire des tumeurs chez les plantes. Un plasmide peut coder, et c’est fréquemment le cas, à la fois pour la résistance aux antibiotiques et une ou plusieurs autres propriétés.Structure et isolementLe plasmide est constitué d’une double hélice d’ADN circulaire surenroulé. Chaque brin est formé d’un enchaînement de quatre bases: adénine (A), guanine (G), thymine (T), cytosine (C) qui se succèdent selon une séquence caractéristique. La séquence des bases d’un brin est complémentaire de la séquence des bases de l’autre brin, A correspondant à T et C à G. La taille des plasmides est très variable, de 1/10 à 1/1 000 de la taille du chromosome bactérien.L’isolement des plasmides peut être réalisé par plusieurs techniques: électrophorèse, ultracentrifugation, microscopie électronique. La microscopie électronique n’est pas un moyen d’isolement mais permet la visualisation et la mesure.Électrophorèse en gel d’agarose d’un lysat bactérienLes bactéries sont lysées, l’ADN est extrait, et un échantillon d’ADN purifié est placé dans un puits à l’une des extrémités de la couche de gel. On fait passer un courant électrique. Les molécules d’ADN plasmidiques migrent vers l’électrode positive à une vitesse inversement proportionnelle au logarithme de leur longueur (fig. 2). Les bandes d’ADN dans le gel sont révélées par un colorant fluorescent qui se fixe électivement sur l’ADN, le bromure d’éthidium, et photographiées en lumière ultraviolette. Cette technique est simple, rapide, permet d’analyser un grand nombre de souches en même temps et a un grand intérêt épidémiologique. Cependant les résultats obtenus par cette technique sont difficilement interprétables lorsque la bactérie héberge plusieurs plasmides de résistance différents, ce qui est fréquent chez les souches résistantes isolées en milieu hospitalier. En effet, chaque plasmide peut donner naissance à plusieurs bandes dans le gel selon sa conformation: structure double brin surenroulée, forme circulaire ouverte par coupure d’un des brins de l’ADN, enfin forme linéaire correspondant à la coupure des deux brins.Ultracentrifugation en chlorure de césium-bromure d’éthidiumIl est possible d’identifier l’ADN plasmidique et de le séparer de l’ADN chromosomique par centrifugation à très grande vitesse dans un gradient de densité de chlorure de césium. L’ADN plasmidique surenroulé est capable d’intercaler entre ses bases moins de bromure d’éthidium que l’ADN chromosomique linéaire. Cette fixation différentielle crée une différence de densité aboutissant au cours de la centrifugation à la formation dans le gradient de deux bandes séparées correspondant l’une à l’ADN chromosomique, l’autre à l’ADN plasmidique (fig. 3). L’ADN plasmidique ainsi purifié peut être conservé et analysé plus avant par microscopie électronique.Microscopie électroniqueL’examen au microscope électronique d’un étalement du contenu plasmidique d’une souche bactérienne permet de déterminer le nombre de plasmides hébergés par cette souche et de mesurer la taille de chacun des plasmides (fig. 1).Critères d’identification et classificationIncompatibilitéDeux plasmides identiques ou étroitement apparentés ne peuvent pas coexister de façon stable dans la même bactérie: ils sont dits incompatibles. À partir d’une bactérie dans laquelle on introduit deux plasmides apparentés, au fur et à mesure de la succession des générations, les bactéries filles se divisent en deux populations qui ne possèdent chacune que l’un ou l’autre plasmide. Au contraire, deux plasmides non apparentés peuvent se maintenir de façon stable dans la même bactérie: ils sont dits compatibles. On isole couramment de bactéries multirésistantes six à huit plasmides différents coexistant dans la même bactérie donc compatibles. On définit ainsi des groupes d’incompatibilité. Deux plasmides incompatibles appartiennent au même groupe d’incompatibilité, et deux plasmides compatibles appartiennent à des groupes d’incompatibilité différents. L’incompatibilité est un phénomène que l’on peut observer dans tous les genres bactériens dont on a étudié les plasmides. Cependant, les plasmides d’entérobactéries sont ceux qui ont été les plus étudiés, on distingue une vingtaine de groupes d’incompatibilité différents chez les plasmides d’entérobactéries autotransférables chez Escherichia coli. Les plasmides du même groupe d’incompatibilité ont des tailles comparables, un degré élevé d’homologie entre leurs ADN (cf. infra ), une spécificité d’hôte voisine. En revanche, des plasmides appartenant à des groupes d’incompatibilité différents peuvent porter des caractères de résistance aux mêmes antibiotiques (cf. chap. Variation ), et il n’existe pas de relation entre groupe d’incompatibilité et caractères de résistance codés par les différents plasmides. Le phénomène d’incompatibilité est étroitement lié à la réplication et à la répartition des plasmides; il semble que des plasmides du même groupe d’incompatibilité possèdent des unités de conduite de réplication identiques, et le même appareil de séparation. Il existerait donc au moins autant d’unités de réplication et de systèmes de répartition que de groupes d’incompatibilité.Spectre de digestion de l’ADN plasmidique par des endonucléases de restrictionLes endonucléases de restriction sont des enzymes d’origine bactérienne qui coupent l’ADN plasmidique en des sites spécifiques, chacune de ces enzymes reconnaissant une séquence de paires de base particulière. La coupure du plasmide par différentes enzymes de restriction aboutit pour chaque enzyme à une série de fragments d’ADN; les fragments obtenus sous l’action d’une endonucléase donnée sont séparés par électrophorèse en gel d’agarose selon leur taille. Chaque sorte de plasmide donne ainsi, après traitement par une endonucléase, un diagramme de coupure caractéristique. Deux plasmides ayant des diagrammes de coupure par plusieurs enzymes superposables appartiennent à des espèces moléculaires voisines ou identiques (fig. 4).HybridationLa parenté entre les ADN de deux plasmides peut être mesurée par hybridation; il peut s’agir d’hybridation entre plasmides entiers ou entre fragments d’ADN plasmidique après coupure par une endonucléase de restriction. L’ADN non radioactif du premier plasmide est fixé soit entier, soit après fragmentation et séparation par électrophorèse, sur un filtre, et l’hybridation se fait avec l’ADN radioactif préparé avec l’autre plasmide. La mesure de la radioactivité fixée sur le filtre se fait soit par comptage de la radioactivité dans le cas des plasmides entiers, soit par autoradiographie pour les ADN fragmentés. Les plasmides appartenant au même groupe d’incompatibilité ont un très fort pourcentage d’homologie ADN-ADN.Analyse d’hétéroduplex en microscopie électroniqueOn peut mesurer l’importance de l’homologie entre les ADN de deux plasmides par l’analyse d’hétéroduplex en microscopie électronique. Un hétéroduplex est le résultat de la réassociation de deux brins d’ADN provenant chacun d’un plasmide différent. Les régions constituées de doubles brins d’ADN correspondent aux régions d’homologie. Les boucles simple brin correspondent aux régions hétérologues. Cette technique permet de détecter et de mesurer l’homologie entre deux plasmides (fig. 5).3. Plasmides R: plasmides de résistance aux antibiotiquesHistoriqueC’est vers la fin des années cinquante que deux microbiologistes japonais, Ochiai et Akiba, observèrent au cours d’une épidémie de dysenterie bacillaire l’apparition de bactéries résistantes à trois antibiotiques à la fois, rendant les malades atteints insensibles au traitement par des antibiotiques habituellement efficaces. Un mécanisme de mutation chromosomique-sélection, décrit par Ledeberg en 1940 et qui permettait d’expliquer les résistances bactériennes observées jusque-là, ne permettait pas d’expliquer cette triple résistance simultanée à trois familles d’antibiotiques différentes. Les auteurs japonais montrèrent alors que cette résistance multiple se transférait entre bactéries intestinales dans le tube digestif des malades, puis ils reproduisirent ce transfert in vitro entre colibacilles et shigelles et démontrèrent que ces bactéries résistantes possèdent en plus de leur chromosome d’autres structures composées d’ADN portant les gènes responsables de cette résistance multiple transférable: ce sont les plasmides de résistance aux antibiotiques ou plasmides R. La découverte de ces éléments génétiques extrachromosomiques mobiles apparut d’emblée d’une importance considérable en clinique par leur incidence sur l’arsenal thérapeutique antibiotique. Les plasmides R permettent aux bactéries d’acquérir la résistance à un ou plusieurs antibiotiques d’emblée; cette résistance est transférable de bactérie à bactérie de la même espèce mais aussi d’espèces voire de genres différents; la résistance peut apparaître ainsi dans une espèce bactérienne jusque-là sensible, la sélection par un antibiotique peut être responsable de la sélection de la résistance à plusieurs antibiotiques non employés, enfin la coexistence possible de plusieurs plasmides R dans une seule cellule bactérienne permet d’expliquer l’émergence dans les milieux à forte pression de sélection antibiotique comme le milieu hospitalier de bactéries multirésistantes responsables d’échec thérapeutique. Depuis leur découverte chez les entérobactéries, des plasmides R ont pu être mis en évidence chez la plupart des bactéries d’intérêt médical, vétérinaire ou industriel.Espèces bactériennes hébergeant des plasmidesDepuis leur découverte chez Shigella dysenteriae , des plasmides de résistance ont été isolés dans la plupart des bactéries pathogènes. Des plasmides ont été mis en évidence chez les bacilles à Gram négatif (entérobactéries diverses, Yersinia , Pseudomonas , Acinetobacter , Aeromonas , Vibrio , Haemophilus , Pasteurella , Bordetella , Campylobacter , Bacteroides fragilis ), les bacilles à Gram positif (Actinomyces , Bacillus , Clostridium perfringens ), les cocci à Gram positif (staphylocoques, streptocoques) et les cocci à Gram négatif (gonocoques). Le pneumocoque et le méningocoque restent les deux seules bactéries de grande importance en pathologie infectieuse dépourvues pour l’instant de plasmides. Des plasmides de résistance peuvent être isolés de nombreuses bactéries de l’environnement, en particulier chez les Streptomyces , bactéries productrices d’antibiotiques. Ces bactéries possèdent les gènes de résistance qui les protègent de l’action de l’antibiotique qu’elles produisent. Ces bactéries productrices d’antibiotiques seraient le réservoir et l’origine de la dissémination des gènes de résistance chez les bactéries pathogènes.DiversificationDeux mécanismes, la mutation et la transposition, permettent la diversification du matériel plasmidique.MutationDes mutations spontanées ou provoquées par des agents mutagènes permettent d’isoler à partir du plasmide originel des variants ou délétants qui sont très utiles pour étudier les propriétés des plasmides: étude de la réplication à l’aide de mutants thermosensibles, étude du transfert avec des mutants déréprimés pour le transfert ou au contraire mutants ayant perdu la transférabilité. Ces mutants ont peu d’incidence sur le phénomène de résistance des antibiotiques.TranspositionLes plasmides peuvent acquérir de nouveaux gènes, en particulier des gènes de résistance aux antibiotiques par le mécanisme de transposition. Certains gènes responsables de la résistance aux antibiotiques sont inclus dans un petit fragment d’ADN qui a la propriété de se détacher ou de «s’exciser» de la molécule d’ADN qui le contient et de s’insérer sur une autre molécule d’ADN. Ces morceaux d’ADN sauteurs ont été appelés «transposons». Certains gènes de résistance sont donc capables par ce mécanisme de sauter d’un plasmide à un autre, d’un plasmide sur un chromosome ou d’un chromosome sur un plasmide. Le phénomène de transposition est un mécanisme qui assure une dissémination très efficace des gènes de résistance. La transposition est un phénomène différent de la recombinaison classique. Elle ne nécessite pas l’enzyme recombinase, elle ne nécessite pas d’homologie entre les ADN donneurs et receveurs du transposon. C’est ainsi que des plasmides appartenant à des groupes d’incompatibilité différents et sans homologie ADN-ADN peuvent porter des gènes de résistance identiques. Les transposons connus mesurent de 2,5 à 20 kilobases, et portent en plus des gènes responsables de la résistance aux antibiotiques, des gènes codant pour des enzymes responsables du mécanisme de transposition et de sa régulation. De nombreux transposons codant pour la résistance à différents antibiotiques ont été caractérisés: 廓-lactamines, streptomycine, kanamycine, gentamicine, tétracycline, érythromycine, chloramphénicol, triméthoprime, sulfamides, phosphomycine. La transposition permet d’expliquer d’une part comment des plasmides R sont capables au cours de leur évolution d’acquérir de nouveaux gènes de résistance; d’autre part elle explique comment des plasmides sans fonction connue et que l’on appelle «cryptiques» peuvent acquérir des gènes de résistance par le mécanisme de transposition et devenir ainsi des plasmides de résistance. La résistance peut apparaître ainsi dans des espèces bactériennes jusque-là sensibles. Ce phénomène a permis d’expliquer l’apparition voici quelques années de souches résistant à la pénicilline chez les Haemophilus et les gonocoques, espèces jusque-là sensibles à cet antibiotique, par la transposition sur des plasmides cryptiques, connus dans ces espèces, de gènes de résistance provenant d’Escherichia coli .Importance pratiqueLes mécanismes de résistance codés par les plasmidesLes plasmides sont capables de rendre les bactéries résistantes par différents mécanismes qui impliquent généralement la synthèse de protéines codées par des gènes plasmidiques: production d’enzymes inactivant l’antibiotique en modifiant sa molécule, modification de la cible de l’antibiotique par une enzyme, diminution de la quantité d’antibiotique transportée à l’intérieur de la bactérie, substitution d’une molécule nouvelle à la place de la cible de l’antibiotique. La majorité des antibiotiques est inactivée par l’un ou l’autre de ces mécanismes, et rares sont les antibiotiques pour lesquels aucune résistance plasmidique n’a été décelée.Outils pour génie génétiqueL’existence de plasmides de résistance n’a pas que des aspects néfastes en amputant l’arsenal thérapeutique. Ce sont des outils particulièrement efficaces dans les expériences de génie génétique. Un des champs d’application privilégiés du génie génétique est l’introduction de gènes codant pour des protéines animales ou humaines dans Escherichia coli pour transformer cette bactérie en usine à fabriquer des protéines d’intérêt thérapeutique ou industriel: hormones, insuline, interféron, etc. Il n’est pas possible actuellement d’introduire directement un fragment d’ADN venant d’organismes supérieurs dans une bactérie et de le fixer sur le chromosome bactérien pour que cette information soit répliquée et transmise à la descendance de la bactérie; un intermédiaire que l’on appelle «vecteur» est indispensable à cette opération. Les plasmides de résistance sont avec les phages les vecteurs essentiellement utilisés. Le plasmide choisi est isolé de la bactérie résistante, purifié, et coupé par une endonucléase de restriction. Le fragment d’ADN étranger peut alors être inséré dans le plasmide ouvert, la molécule hybride recircularisée (fragment d’ADN + plasmide) est introduite chez Escherichia coli. Les bactéries ayant reçu la molécule hybride sont aisément sélectionnées par leur résistance à un antibiotique, codée par le plasmide. Introduire un gène étranger par cet artifice chez Escherichia coli est une opération simple aujourd’hui. Cependant, l’expression de ce gène «importé» dans un environnement qui lui est totalement étranger reste largement imprévisible. Ce phénomène est à l’origine des échecs relativement nombreux rencontrés par les précurseurs dans ce domaine, difficultés qui sont actuellement partiellement résolues.Problèmes épidémiologiquesActuellement, la résistance d’origine plasmidique rend compte de plus de 80 p. 100 de la résistance bactérienne aux antibiotiques. Dans la nature, chez l’homme sain ou malade, et chez les animaux, les bactéries porteuses de plasmides de résistance sont de plus en plus nombreuses. Les quantités considérables d’antibiotiques déversées dans l’environnement à des fins thérapeutiques ou autres depuis quarante ans sont vraisemblablement à l’origine de la sélection de bactéries résistantes. L’antibiosupplémentation de l’alimentation du bétail, en particulier, a été mise en cause dans l’augmentation de fréquence des bactéries multirésistantes. Ainsi, on a pu montré que 100 p. 100 des porcs et des veaux recevant des antibiotiques hébergeaient dans leur tube digestif des entérobactéries porteuses de plasmides de résistance transférables. Chez les éleveurs ne recevant pas d’antibiotiques, mais en contact avec les animaux, le portage digestif d’entérobactéries résistantes est de 50 p. 100 ou plus; en revanche, l’étude d’une population isolée de Bornéo sans contact avec les antibiotiques a montré que moins de 1 p. 100 des bactéries des selles pouvaient transférer leur résistance. Dans l’environnement, on peut isoler des bactéries multirésistantes dans les égouts, sur les plages par exemple. On peut donc supposer que c’est l’utilisation massive des antibiotiques qui est responsable d’un si brutal changement écologique. La dissémination de la résistance s’effectue par plusieurs mécanismes parfois étroitement intriqués. Une souche d’une bactérie pathogène peut acquérir un plasmide de résistance provenant d’une entérobactérie du tube digestif, puis cette souche disséminer dans la population (ou de malade à malade), réalisant de la sorte une épidémie due à une seule et unique souche. Il existe de nombreux exemples de telles épidémies: épidémie de fièvre typhoïde en Amérique latine due à une Salmonella porteuse d’un plasmide de résistance au chloramphénicol et à l’ampicilline, épidémies hospitalières à bactéries multirésistantes responsables d’infections nosocomiales dans un milieu à forte pression de sélection par les antibiotiques. En milieu hospitalier, on peut suivre un autre mode de dissémination de la résistance: des épidémies de plasmides, le même plasmide de résistance se propageant au fil du temps dans plusieurs espèces bactériennes différentes dans le même hôpital. De telles épidémies de plasmides de résistance à la gentamicine et au triméthoprime ont été bien individualisées par la comparaison fine des plasmides isolés des différentes espèces de bactéries résistant aux mêmes antibiotiques. Enfin, plus récemment, on a pu démontrer l’identité des gènes de résistance et isoler des transposons identiques sur différents plasmides hébergés par des espèces ou des genres bactériens très divers. Il s’agit alors d’épidémies de transposons. Une appréciation correcte du danger de la résistance plasmidique repose sur l’épidémiologie des plasmides et des gènes de résistance permettant de connaître, de suivre, éventuellement de prévoir et d’empêcher leur dissémination.La détection de nouvelles résistances et la surveillance de l’évolution des résistances connues nécessitent une étroite collaboration entre les thérapeutes, les microbiologistes, les épidémiologistes et les biologistes cellulaires. Enfin, il existe un lien indiscutable entre la résistance des bactéries et l’utilisation des antibiotiques dans un hôpital; une meilleure connaissance de l’écologie locale de la résistance et des différences notables qui existent dans le degré du pouvoir sélectionnant de chaque antibiotique devrait conduire à des modifications dans les habitudes de prescription et à une diminution de la résistance.4. Facteurs sexuels bactériensLe facteur sexuel F d’Escherichia coli est un épisome dont la taille est d’environ 1 p. 100 de celle du chromosome et dont la propriété essentielle est de rendre «fertiles» les bactéries qui le portent. Cette fertilité se manifeste de différentes manières suivant que le facteur sexuel est autonome (bactérie +) ou intégré dans le chromosome (bactérie Hfr). Les bactéries + ou Hfr sont dites donatrices ou mâles, alors que les bactéries dépourvues de facteur sexuel (F size=1漣) sont dites réceptrices ou femelles.La recombinaison par conjugaison bactérienneLa découverte du facteur F est liée à l’étude de la recombinaison génétique chez Escherichia coli K12. En 1946, Lederberg et Tatum montraient que, en mélangeant deux cultures bactériennes X (abc DEF) et Y (ABC def ) respectivement polyauxotrophes, c’est-à-dire exigeantes en produits variés (abc pour X, et def pour Y), on récupérait des bactéries prototrophes Z (ABCDEF) ne nécessitant aucun de ces produits. Les bactéries Z, ne pouvant être des mutants réverses de X ou Y en raison du changement simultané de plusieurs caractères, sont des «recombinants» entre X et Y. Rapidement, on s’aperçut que les bactéries X et Y ne jouaient pas un rôle identique. La survie d’une des deux bactéries X ou Y est nécessaire à la production de recombinants, alors que l’on peut tuer l’autre avant ou pendant le croisement sans diminuer le nombre de recombinants qui apparaissent. La bactérie Y, par exemple, dont la survie est absolument nécessaire, est la bactérie réceptrice: en elle a lieu le processus de recombinaison au niveau moléculaire. La bactérie X est alors la bactérie donatrice (F+). À partir des bactéries +, on a pu isoler des variants qui ne donnent plus de recombinants avec une souche F size=1漣. Par ailleurs, on peut classer les souches d’Escherichia coli en deux types: les souches qui, croisées entre elles, ne donnent jamais de recombinants (souche F size=1漣) et les souches qui donnent des recombinants avec n’importe quelle autre souche (souche fertile +).Les types sexuels + et F size=1漣Le caractère + ou F size=1漣 qui donne aux bactéries le phénotype donneur ou receveur apparaissait donc jusqu’en 1953 comme un caractère génétique classique, déterminé par le chromosome et qui pouvait muter par exemple de + vers F size=1漣. Cependant, certaines propriétés très particulières du caractère + ont montré qu’il n’était pas codé par des gènes chromosomiques. Lors d’un croisement entre une souche + et une souche F size=1漣, on obtenait de l’ordre de 10 size=1漣6 à 10 size=1漣7 recombinant pour les caractères chromosomiques; en revanche, la majorité des bactéries réceptrices sont converties en bactéries +. C’était la première preuve d’indépendance du facteur F par rapport au chromosome. Cette indépendance devait se confirmer par le fait que, lorsqu’on mélange un nombre connu de bactéries donatrices avec un nombre plus grand de bactéries F size=1漣, ces dernières sont converties en bactéries + à une vitesse supérieure à celle de la multiplication bactérienne. Cela signifie que le facteur F se réplique de manière autonome et à une vitesse qui peut être supérieure à celle du chromosome, ce qui lui permet d’envahir par transfert toute une culture: on dit qu’il est «infectieux» (fig. 6). Enfin, le facteur F peut être spontanément perdu par une souche, à un taux qui est en général beaucoup plus grand que le taux de mutation. F est donc un élément indépendant du chromosome, capable de se répliquer à une vitesse propre et de se transférer de bactérie à bactérie par conjugaison à haute fréquence. C’est un plasmide autotransférable.Les mutants HfrÀ partir de bactéries +, Cavalli-Sforza et Hayes ont isolé des mutants qui produisaient mille fois plus de recombinants lorsqu’on les croisait avec des bactéries F size=1漣. Ces mutants se comportaient comme des «supermâles»: on les a appelés souches Hfr (haute fréquence de recombinaison). En plus du problème de la fréquence des recombinants, les croisements Hfr 憐 F size=1漣 présentaient essentiellement deux caractères qui les distinguaient des croisements + 憐 F size=1漣. Les croisements + 憐 F size=1漣 conduisent à un nombre faible mais constant de recombinants Z (ABCDEF), quel que soit le caractère sélectionné. Cela montre donc que les bactéries donatrices + peuvent transférer n’importe quel segment de leur chromosome avec une égale fréquence. Au contraire, dans un croisement Hfr 憐F size=1漣, le nombre de recombinants formés est très inégal suivant le caractère sélectionné. Par ailleurs, sauf cas exceptionnel, les bactéries réceptrices ne sont pas transformées en bactéries + dans un croisement Hfr 憐 F size=1漣. Ces propriétés des donneurs Hfr ont suggéré que, dans ces bactéries, le facteur sexuel F existe sous un état différent de celui sous lequel il existe dans des bactéries +. Il a été démontré par la suite que ce second état du facteur F est l’état intégré au chromosome bactérien où l’ADN de F est en continuité avec l’ADN de la cellule hôte (fig. 7). Nous avons déjà signalé qu’on a donné le nom d’épisome à de tels éléments génétiques capables d’exister sous deux états. Cette généralisation, principalement due à Jacob et Wollman, est née de l’étude du facteur F mais aussi des bactériophages tempérés et des facteurs colicinogènes.Équilibre réplicatif et notion de répliconJacob, Brenner et Cuzin ont proposé d’appeler «réplicon» les structures génétiques dont la réplication autonome est indépendante de celle du chromosome. Un réplicon est donc une unité de réplication. Dans cette hypothèse, que des travaux ultérieurs ont partiellement confirmée, un réplicon a au moins deux types de fonctions et comprend deux sortes de matériel génétique au sens fonctionnel.Le premier type comporte les fonctions obligatoires intervenant spécifiquement dans la réplication et permettant à la molécule d’ADN qui les porte de se répliquer avec une vitesse et une régulation caractéristiques de ce replicon. Les déterminants génétiques impliqués dans cette fonction seraient au moins deux. Il existerait un gène de structure codant pour la synthèse d’une protéine dite initiatrice qui agirait sur un second gène, le réplicateur, «véritable promoteur de la réplication». Lorsque la protéine initiatrice agit sur le réplicateur, la réplication est amorcée et se poursuit tout le long de la molécule d’ADN adjacente au réplicateur (position cis). Ces deux gènes sont absolument spécifiques et l’initiateur produit par un réplicon donné, par exemple un phage, reconnaît le réplicateur de ce phage, mais pas le réplicateur du chromosome bactérien ou d’un autre type de phage.Le second type de fonction est variable et caractériel le type de matériel génétique que l’on étudie: le facteur F porte des gènes qui permettent un transfert de matériel génétique; les facteurs de résistance (cf. chap. 3) codent pour des enzymes qui détruisent certains antibiotiques; le chromosome bactérien contient toute l’information nécessaire à la multiplication de la bactérie. Ces gènes se répliquent à la vitesse du réplicon qui les porte.On sait actuellement que le facteur F se multiplie indépendamment du chromosome mais en accord avec lui de façon à conserver le rapport de un épisome par chromosome. Cependant, il peut se produire des cas où cet équilibre est rompu; par exemple, si des bactéries + sont mises en contact avec des bactéries F size=1漣, le facteur F envahit la culture par le biais du transfert (cf. chap. 2, Le Transfert ). Par ailleurs, certaines bactéries peuvent perdre le facteur F et deviennent F size=1漣. La fréquence de cette perte est de l’ordre de 10 size=1漣2 à 10 size=1漣4 selon les souches; elle peut être accrue si on traite les bactéries par des colorants comme l’acridine orange. Notons que la transition spontanée du caractère + vers F size=1漣 est possible alors que le passage de l’état F size=1漣 vers l’état + nécessite le contact avec une souche +.La réplication du facteur F a été étudiée en utilisant des mutants thermosensibles. Dans ces expériences, on utilise en général des facteurs F (cf. infra ) qui portent des marqueurs génétiques faciles à repérer comme le caractère lac+ (utilisation du lactose). Si le facteur F size=1漣 lac+ est étudié dans une bactérie lac size=1漣 (incapable d’utiliser le lactose), le caractère lac+ sera un témoin de la présence du facteur F . Il a été obtenu des mutants thermosensibles de F lac+ que l’on désigne Fts-lac+. Dans ces bactéries, le Fts-lac+ se réplique à basse température (25 0C), mais à haute température (42 0C) les bactéries deviennent lac size=1漣 car le Fts-lac+ ne se multiplie pas. À 42 0C, on trouve cependant quelques clones lac+: dans ces bactéries, le Fts-lac+ a donc pu se perpétuer. Une étude approfondie de ces bactéries a montré qu’elles ont conservé l’épisome dont la réplication est thermosensible, mais que celui-ci s’est intégré dans le chromosome, et est donc répliqué par le «réplicon chromosome». Chez ces mutants, le chromosome «sauve» l’épisome de l’effet de la température. De manière réciproque, des mutants de chromosomes bactériens thermosensibles pour la réplication peuvent être sauvés par l’intégration d’un facteur F normal. C’est ici le facteur F qui prend la réplication du chromosome sous son contrôle. L’ensemble de ces expériences illustre précisément les prévisions que l’on pouvait faire à partir de la théorie du réplicon.Propriétés des bactéries +Les propriétés particulières qui différencient les bactéries portant le facteur F des bactéries F size=1漣 dépourvues de ce facteur sont liées à l’expression de la fonction de transfert du facteur sexuel. Tout d’abord, les bactéries + présentent des différences morphologiques liées essentiellement à l’existence à leur surface d’un petit nombre de «poils» spécifiques appelés «F pili». La synthèse des F pili est constante chez les bactéries +, alors que la synthèse d’éléments semblables est soumise à régulation chez des bactéries portant des facteurs de résistance. Si l’on enlève les F pili par agitation de la culture dans un mixer, ou si l’on utilise un mutant dénué de pili, le transfert génétique du facteur F n’a pas lieu. Comme ils sont creux, ces «poils» sont peut-être des organes de copulation à travers lesquels s’effectue le transfert de matériel génétique. Ce point a été discuté car certains auteurs estiment que les pili sont simplement des «câbles d’amarrage» servant durant la période initiale de la conjugaison à l’appariement des bactéries. L’existence de ces poils explique un certain nombre de différences biochimiques et immunologiques entre les bactéries donatrices et réceptrices. Par ailleurs, ces F pili portent les récepteurs de bactériophages qui ne se fixent et ne se multiplient que chez les bactéries mâles.Sur le plan biochimique, on peut retrouver dans le cytoplasme des bactéries + des molécules d’ADN circulaire ayant sensiblement la même composition en bases nucléiques que l’ADN du chromosome d’Escherichia coli (50 p. 100 de guanine-cytosine).Au niveau génétique, la propriété de transfert doit donc impliquer l’existence de deux types de gènes: ceux qui conduisent à la synthèse des F pili et ceux qui permettent à la bactérie donatrice d’injecter le facteur F à la bactérie réceptrice tout en restant elle-même +. Des mutants du facteur F ont été isolés, qui sont incapables d’assurer leur propre transfert et le transfert de gènes chromosomiques. La plupart de ces mutants sont incapables de synthétiser des F pili; les autres seraient incapables d’injecter l’ADN dans les bactéries F size=1漣. Il semble y avoir indépendance entre la réplication du facteur F et le transfert de l’ADN entre bactéries conjugantes. Lorsque le facteur F est normal, les bactéries + transmettent le facteur F avec une fréquence élevée et les gènes chromosomiques avec une fréquence faible. On admet que le transfert à basse fréquence des gènes chromosomiques est dû à l’existence dans les cultures de souches + de rares variants Hfr.Propriétés des bactéries Hfr.Les bactéries Hfr présentent les mêmes caractères morphologiques que les bactéries +. Mais les bactéries expriment la fonction de transfert d’une façon différente, liée au fait que le facteur F est intégré dans le chromosome bactérien. Dans ce cas, F est répliqué passivement comme un segment d’ADN chromosomique, mais en revanche «impose» la fonction de transfert à tout le chromosome bactérien. Cela se traduit par les caractères particuliers que présente la conjugaison entre une bactérie Hfr et une bactérie F size=1漣: ces croisements produisent beaucoup plus de recombinants qu’un croisement + 憐 F size=1漣; la proportion des recombinants est très variable suivant le caractère chromosomique sélectionné (de 10 size=1漣3 à 10 size=1漣4); la nature des marqueurs transmis à haute fréquence varie avec l’origine de la source Hfr utilisée. En ce qui concerne le facteur sexuel, il n’est pas infectieux dans les souches Hfr, et il est très rarement transmis lors du processus de conjugaison. Jacob et Wollman ont démontré que le transfert des gènes chromosomiques est orienté et progressif. Ces conclusions reposent sur une étude cinétique du processus dans des expériences de conjugaison interrompue.On peut donner actuellement une explication assez claire du comportement des bactéries Hfr. Les bactéries Hfr sont des mutants de bactéries + dans lesquelles le facteur F s’est intégré dans le chromosome. Le facteur F est alors capable de promouvoir le transfert de gènes chromosomiques. Le lieu d’insertion du facteur F dans ce chromosome détermine quels seront les marqueurs transmis en premier ou en dernier à partir d’une bactérie Hfr donnée (fig. 8). Le nombre de points d’insertion est élevé mais limité, aussi existe-t-il un nombre limité de types d’Hfr. D’autre part, le chromosome bactérien étant cent fois plus long que le facteur F, le transfert du chromosome est long, peut s’interrompre spontanément par cassure de ce chromosome ou dissociation des paires de bactéries. Cela explique que les gènes proches de la «tête» du chromosome de l’Hfr entreront souvent dans la femelle et donneront de nombreux recombinants, alors que les gènes situés en «queue» seront souvent non transférés. D’autre part, dans les souches Hfr, la réplication du facteur F est liée à celle du chromosome, et ce F ne peut donc être infectieux, comme c’est le cas dans les souches +.L’état Hfr est assez instable, et on peut revenir vers l’état + par excision du facteur F. Lors de ce processus, il peut se produire diverses anomalies. Cela conduit alors à la formation d’épisomes portant des fragments de chromosome (fig. 9): ce sont les facteurs F déjà cités. Ils conservent en général toutes les caractéristiques de F, mais emmènent des segments d’ADN bactériens de tailles variables, pouvant atteindre 10 p. 100 du chromosome. Des F peuvent donc transporter des gènes bactériens d’une souche à une autre. Ce phénomène s’appelle la sexduction. Les facteurs F sont très utilisés en génétique bactérienne soit en tant que marqueurs des facteurs F (cf. supra ), soit pour construire des diploïdes partiels (mérodiploïdes). Les bactéries sont en effet haploïdes; et, pour étudier le caractère dominant ou récessif d’une mutation, il faut avoir recours à des artifices génétiques. Les facteurs F et F sont donc des outils très utiles en génétique moléculaire.
Encyclopédie Universelle. 2012.
